Zum Erfolg der Translationalen Lungenforschung leistet hochmodernes Biobanking von Lungengewebe, Blut und anderen Biomaterialien einen wichtigen Beitrag. In der Heidelberger Lungenbiobank werden aktuell ca. 8000 frisch-gefrorene Gewebeproben für Forschungszwecke aufbewahrt.
Dr. Thomas Muley, Leiter der Heidelberger Lungenbiobank, war von 2012-2019 einer der Koordinatoren der Plattform Biobank des Deutschen Zentrums für Lungenforschung (DZL), welche eine Reihe von translationalen Forschungsprojekten als zentrale Anlaufstelle für den vereinfachten Zugriff auf dezentral gelagerte Biomaterialien über die verschiedenen DZL Standorte hinweg unterstützt.
Die Lungenbiobank der Thoraxklinik am Universitätsklinikum Heidelberg ist eine akkreditierte Außenstelle (DAP-IS-4153.04 entspricht DIN EN ISO / IEC 17020) des Nationalen Centrums für Tumorerkrankungen Heidelberg (NCT) und integriert in die BioMaterialBank Heidelberg (BMBH). Die NCT Gewebebank ist eine eingetragene Biobank im Register der Technologie- und Methodenplattform für die Forschung (TMF) der German Biobank Alliance (GBA) und im europäischen Biobankenregister (BBMRI-ERIC)
Kontakt:
Dr. Thomas Muley
Sektion Translationale Forschung (STF)
Thoraxklinik am Universitätsklinikum Heidelberg
Röntgenstraße 1
69126 Heidelberg
Germany
Tel. +49 (0) 6221 396 1110
Fax +49 (0) 6221 396 1652
Die Heidelberger Lungenbiobank lagert bzw. stellt die folgenden Biomaterialien für Diagnose und Forschungszwecke bereit:
Cryo-präserviertes Tumor- und korrespondierendes Lungengewebe
FFPE-Gewebe (via NCT-Gewebebank)
Multi-Tissue-Arrays (via NCT-Gewebebank)
Blutproben (Serum, Plasma, Buffy Coat), PAXGene
„Epithelial lining fluid“ der Atemwege
Extrahierte Nukleinsäuren
Primäre Zellkulturen (inkl. Spheroide)
Ausatemkondensat (Exhalted Breath Condensate)
Broncho-alveoläre Lavage und induziertes Sputum
Gewebeproben
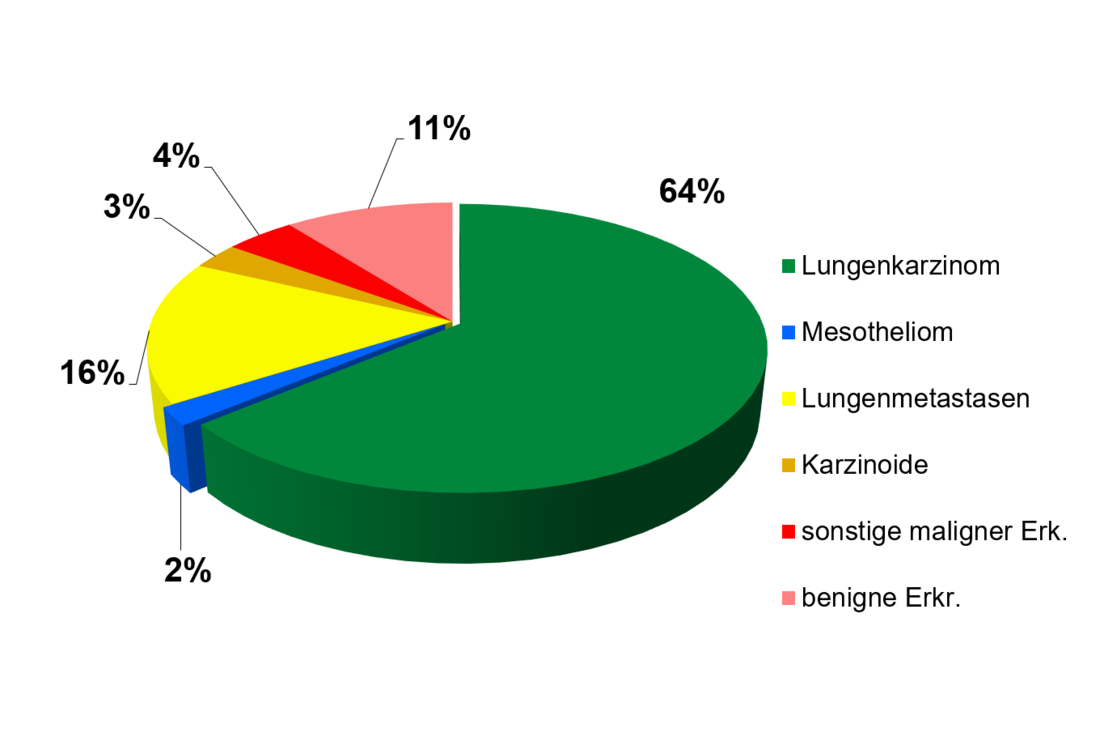
Im Rahmen des Biobanking werden jedes Jahr in etwa 400-500 neue Gewebeproben und 3000 - 4000 neue Blutproben gesammelt und aufgearbeitet. Ungefähr zwei Drittel der Gewebeproben stammen von Lungekrebspatienten (Tumorgewebe mit korrespondierendem tumorfreien Lungengewebe, Lymphknoten). Des Weiteren werden Gewebe von anderen bösartigen primären Thoraxerkrankungen (Karzinoid, Mesotheliom, Thymom, Lungen Sarkom), sowie Lungenmetastasen -gewebe extrathorakaler Tumorerkrankungen (Kolorektales Karzinom, Nieren Karzinom etc.) und in geringerem Umfang auch Gewebe von gutartigen Lungenerkrankungen gesammelt (Hamartome, Interstitielle Lungenerkrankungen, COPD, Pneumothorax) (Abb.1).
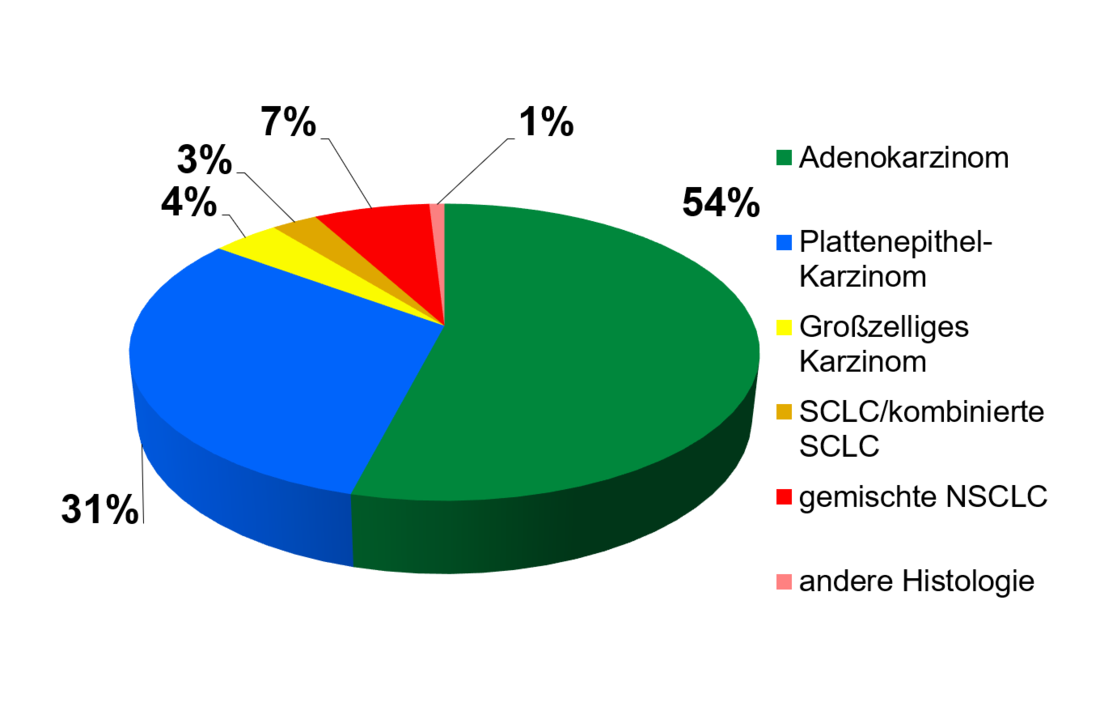
Die häufigste Form des Bronchialkarzinoms ist der sogenannte nicht-kleinzellige Lungenkrebs. Abgekürzt wird diese Form eines Bronchialkarzinoms auch als NSCLC, vom englischen "non-small cell lung cancer". Die NSCLC Tumore sind bekanntermaßen sehr unterschiedlich in Bezug auf beteiligte Tumorzelltypen, der Zusammensetzung des umgebenden Gewebes sowie dem Anteil von Bindegewebe und des Grades der Fibrose und Nekrose. Unser Ziel ist es für die Forschung standardisiert aufgearbeitete Biomaterialien von höchster Qualität zur Verfügung zu stellen. Im diesem Sinne unterliegen unsere Probenaufbereitungsverfahren der Akkreditierung nach DIN EN ISO/IEC 17020.
Standardisierte Arbeitsanweisungen (Standard Operating Procedures, SOPs) werden unter anderem für folgende Probenaufbereitungen angewandt:
- Prozessierung des Gewebes für die Isolierung von Nukleinsäuren
- Isolierung qualitativ hochwertiger Nukleinsäuren (DNA, Gesamt RNA inklusive miRNA)
- Isolierung und Kultivierung von Primärzellen vom Tumorgewebe und tumor-freien Lungengewebe
Die Heidelberger Lungenbiobank verfügt über eine langjährige Expertise einer qualitativ hochwertigen Tumordokumentation, die jedes Jahr ca. 1100 neue Fälle umfasst. Die Lungenbiobank operiert auf Basis eines qualitätsgesicherten Datenmanagement Systems, das im Wesentlichen in das klinische IT System eingegliedert ist.
Ein systemmedizinischer Ansatz zur Stratifizierung von Krebsrezidiven (SMART-CARE) - BMBF-Projekt
Das Grundprinzip von SMART-CARE ist zweifacher Natur: Erstens, der Aufbau einer robusten, auf Massenspektrometrie (MS) basierenden Pipeline, die alle Schritte von der Probensammlung, Analytextraktion und Verarbeitung durch MS in SOPs standardisiert, um eine reproduzierbare Proteom- und Metabolomanalyse zu gewährleisten. Zweitens soll die etablierte Pipeline in einem systemmedizinischen Ansatz zur Vorhersage von Tumorrezidiven eingesetzt werden.
Heidelberger Zentrum für Personalisierte Onkologie (DKFZ-HIPO) Das vom DKFZ etablierte Heidelberger Zentrum für Personalisierte Onkologie bietet eine dezidierte Plattform zur Integration von Genomik, Proteomik und Bioinformatik, die vor allem zur Exzellenz der grundlagenwissenschaftlichen und translationalen Forschung beiträgt.
Forschungsprogramm der Präzisionsonkologie am Nationalen Centrum für Tumorerkrankungen (NCT-POP). Dieses Forschungsprogramm hat zum Ziel individuelle Veränderungsmuster eines jeden Krebspatienten am NCT auszuwerten und die verfügbaren Wirkstoffe und Therapien möglichst optimal für die Behandlung zu nutzen.
Der Krebs Genom Atlas (engl. TCGA) ist eine umfassende Bibliothek sämtlicher Gendefekte bei allen bekannten Krebsarten. Dafür werden seit 2006 durch die Anwendung modernster Genom Analysetechniken und umfassende Sequenzierung Daten erhoben.
Lungscape ist ein translationales Forschungsprojekt der europäischen Thoraxonkologie Pattform (ETOP). Das Programm fokussiert sich auf die molekulare Epidemiologie des Lungenkrebs.
Die NCT Gewebebank eine zentrale Ressource und Technologieplattform für den Heidelberger Campus, gewährt Zugang zu gefrorenen und Parafin-konservierten Tumorgewebe und korrespondierendem tumor-freien Gewebe, den relevanten klinischen Daten, hochmoderne Gewebeaufbereitungstechnologien und umfassendes Projektmanagement.
Technologie- und Methodenplattform für die vernetzte medizinische Forschung e.V. (TMF) ist eine Dachorganisation für die vernetzte medizinische Forschung in Deutschland, die das Deutsche Biobank Register koordiniert. Biobanken spielen eine zentrale Rolle in der modernen biomedizinischen Forschung.
Biobanking and Biomolecular Resources Research Infrastructure (BBMRI) - das europäische Biobankenregister ist ein 54 Mitglieder umfassendes Konsortium, mit mehr als 225 assoziierten Organisationen (hauptsächlich Biobanken) aus mehr als 30 europäischen Ländern, und damit eines der größten Infrastruktur-Projekte Europas.
Leitender Wissenschaftler
Dr. sc. hum. Thomas Muley Leiter der Lungenbiobank Heidelberg, Sektion Translationale Forschung, Thoraxklinik am Universitätsklinikum Heidelberg, Co-Koordinator der DZL-Biobank Plattform
Weitere Wissenschaftler
Dr. Marc A. Schneider, Biobankmanager Lungenbiobank, Sektion Translationale Forschung, Thoraxklinik am Universitätsklinikum Heidelberg
Dr. rer. nat. Michael Andres Meister, Sektion Translationale Forschung, Thoraxklinik am Universitätsklinikum Heidelberg
Dipl. Karsten Senghas, TLRC-Datenmanager, Sektion Translationale Forschung, Thoraxklinik am Universitätsklinikum
Dr. Kadriya Yuskaeva, Datenmanagerin, Sektion Translationale Forschung, Thoraxklinik am Universitätsklinikum Heidelberg
Dr. Mark Kriegsmann, Sektion Thoraxpathologie, Institut für Pathologie, Universitätsklinikum Heidelberg
2020
- Mark Kriegsmann*, Christian Haag, Cleo-Aron Weis, Georg Steinbuss, Arne Warth, Christiane Zgorzelski, Thomas Muley, Hauke Winter, Martin E. Eichhorn, Florian Eichhorn, Joerg Kriegsmann, Petros Christopolous, Michael Thomas, Mathias Witzens-Harig, Peter Sinn, Moritz von Winterfeld, Claus Peter Heussel, Felix J. F. Herth, Frederick Klauschen, Albrecht Stenzinger and Katharina Kriegsmann Deep Learning for the Classification of Small-Cell and Non-Small-Cell Lung Cancer Cancers 2020, 12, 1604; doi:10.3390/cancers12061604
- Shen Y, Wang X, Lu J, Salfenmoser M, Wirsik NM, Schleussner N, Imle A, Freire Valls A, Radhakrishnan P, Liang J, Wang G, Muley T, Schneider M, Ruiz de Almodovar C, Diz-Muñoz A, Schmidt T. Reduction of Liver Metastasis Stiffness Improves Response to Bevacizumab in Metastatic Colorectal Cancer. Cancer Cell. 2020 Jun 8;37(6):800-817.e7. doi: 10.1016/j.ccell.2020.05.005.Cancer Cell. 2020. PMID: 32516590
- Ji X, Mukherjee S, Landi MT, Bosse Y, Joubert P, Zhu D, Gorlov I, Xiao X, Han Y, Gorlova O, Hung RJ, Brhane Y, Carreras-Torres R, Christiani DC, Caporaso N, Johansson M, Liu G, Bojesen SE, Le Marchand L, Albanes D, Bickeböller H, Aldrich MC, Bush WS, Tardon A, Rennert G, Chen C, Byun J, Dragnev KH, Field JK, Kiemeney LF, Lazarus P, Zienolddiny S, Lam S, Schabath MB, Andrew AS, Bertazzi PA, Pesatori AC, Diao N, Su L, Song L, Zhang R, Leighl N, Johansen JS, Mellemgaard A, Saliba W, Haiman C, Wilkens L, Fernandez-Somoano A, Fernandez-Tardon G, Heijden EHFMV, Kim JH, Davies MPA, Marcus MW, Brunnström H, Manjer J, Melander O, Muller DC, Overvad K, Trichopoulou A, Tumino R, Goodman GE, Cox A, Taylor F, Woll P, Wichmann E, Muley T, Risch A, Rosenberger A, Grankvist K, Johansson M, Shepherd F, Tsao MS, Arnold SM, Haura EB, Bolca C, Holcatova I, Janout V, Kontic M, Lissowska J, Mukeria A, Ognjanovic S, Orlowski TM, Scelo G, Swiatkowska B, Zaridze D, Bakke P, Skaug V, Butler LM, Offit K, Srinivasan P, Bandlamudi C, Hellmann MD, Solit DB, Robson ME, Rudin CM, Stadler ZK, Taylor BS, Berger MF, Houlston R, McLaughlin J, Stevens V, Nickle DC, Obeidat M, Timens W, Artigas MS, Shete S, Brenner H, Chanock S, Brennan P, McKay JD, Amos CI. Protein-altering germline mutations implicate novel genes related to lung cancer development Nat Commun. 2020 May 11;11(1):2220. doi: 10.1038/s41467-020-15905-6.
- Rheinheimer S, Heussel CP, Mayer P, Gaissmaier L, Bozorgmehr F, Winter H, Herth FJ, Muley T, Liersch S, Bischoff H, Kriegsmann M, El Shafie RA, Stenzinger A, Thomas M, Kauczor HU, Christopoulos P. Oligoprogressive Non-Small-Cell Lung Cancer under Treatment with PD-(L)1 Inhibitors. Cancers (Basel). 2020 Apr 23;12(4):1046. doi: 10.3390/cancers12041046.
- Duerr J, Leitz DHW, Szczygiel M, Dvornikov D, Fraumann SG, Kreutz C, Zadora PK, Seyhan Agircan A, Konietzke P, Engelmann TA, Hegermann J, Mulugeta S, Kawabe H, Knudsen L, Ochs M, Rotin D, Muley T, Kreuter M, Herth FJF, Wielpütz MO, Beers MF, Klingmüller U, Mall MA. Conditional deletion of Nedd4-2 in lung epithelial cells causes progressive pulmonary fibrosis in adult mice. Nat Commun. 2020 Apr 24;11(1):2012. doi: 10.1038/s41467-020-15743-6.
- Janke F, Bozorgmehr F, Wrenger S, Dietz S, Heussel CP, Heussel G, Silva CF, Rheinheimer S, Feisst M, Thomas M, Golpon H, Günther A, Sültmann H, Muley T, Janciauskiene S, Meister M, Schneider MA. Novel Liquid Biomarker Panels for A Very Early Response Capturing of NSCLC Therapies in Advanced Stages.Cancers (Basel). 2020 Apr 12;12(4). pii: E954. doi: 10.3390/cancers12040954.
- Lukassen S, Chua RL, Trefzer T, Kahn NC, Schneider MA, Muley T, Winter H, Meister M, Veith C, Boots AW, Hennig BP, Kreuter M, Conrad C, Eils R. SARS-CoV-2 receptor ACE2 and TMPRSS2 are primarily expressed in bronchial transient secretory cells. EMBO J. 2020 Apr 14:e105114. doi: 10.15252/embj.2020105114. [Epub ahead of print
- von Eiff D, Bozorgmehr F, Chung I, Bernhardt D, Rieken S, Liersch S, Muley T, Kobinger S, Thomas M, Christopoulos P, Steins M. Paclitaxel for treatment of advanced small cell lung cancer (SCLC): a retrospective study of 185 patients. J Thorac Dis. 2020 Mar;12(3):782-793. doi: 10.21037/jtd.2019.12.74
- Kim-Wanner SZ, Assenov Y, Nair MB, Weichenhan D, Benner A, Becker N, Landwehr K, Kuner R, Sültmann H, Esteller M, Koch I, Lindner M, Meister M, Thomas M, Bieg M, Klingmueller U, Schlesner M, Warth A, Brors B, Seifried E, Bonig H, Plass C, Risch A, Muley T. Genome-wide DNA methylation profiling in early stage I lung adenocarcinoma reveals predictive aberrant methylation in the promoter region of the long non-coding RNA PLUT - an exploratory study.J Thorac Oncol. 2020 Apr 6. pii: S1556-0864(20)30287-2. doi: 10.1016/j.jtho.2020.03.023.
- Kriegsmann K, Zgorzelski C, Kazdal D, Cremer M, Muley T, Winter H, Longuespée R, Kriegsmann J, Warth A, Kriegsmann M. Insulinoma-associated Protein 1 (INSM1) in Thoracic Tumors is Less Sensitive but More Specific Compared With Synaptophysin, Chromogranin A, and CD56. Appl Immunohistochem Mol Morphol. 2020 Mar;28(3):237-242. doi:
- Erber R, Warth A, Muley T, Hartmann A, Herpel E, Agaimy A. BAP1 Loss is a Useful Adjunct to Distinguish Malignant Mesothelioma Including the Adenomatoid-like Variant From Benign Adenomatoid Tumors. Appl Immunohistochem Mol Morphol. 2020: 28: 67-73 doi: 10.1097/PAI.0000000000000700.
2019
- Thunnissen E, Kerr KM, Dafni U, Bubendorf L, Finn SP, Soltermann A, Biernat W, Cheney R, Verbeken E, Warth A, Marchetti A, Speel EM, Pokharel S, Quinn AM, Monkhorst K, Navarro A, Madsen LB, Tsourti Z, Geiger T, Kammler R, Peters S, Stahel RA; European Thoracic Oncology Platform Lungscape Consortium. Programmed death-ligand 1 expression influenced by tissue sample size. Scoring based on tissue microarrays' and cross-validation with resections, in patients with, stage I-III, non-small cell lung carcinoma of the European Thoracic Oncology Platform Lungscape cohort. Mod Pathol. 2020, 33,:792-801 19 Nov 18. doi: 10.1038/s41379-019-0383-9. [Epub ahead of print]
- Serçin Ö, Reither S, Roidos P, Ballin N, Palikyras S, Baginska A, Rein K, Llamazares M, Halavatyi A, Winter H, Muley T, Jurkowska RZ, Abdollahi A, Zenke FT, Neumann B, Mardin BR. A solid-phase transfection platform for arrayed CRISPR screens. Mol Syst Biol. 2019 Dec;15(12):e8983. doi: 10.15252/msb.20198983.
- Dietz S, Christopoulos P, Gu L, Volckmar AL, Endris V, Yuan Z, Ogrodnik SJ, Zemojtel T, Heussel CP, Schneider MA, Meister M, Muley T, Reck M, Schlesner M, Thomas M, Stenzinger A, Sültmann H.Serial liquid biopsies for detection of treatment failure and profiling of resistance mechanisms in KLC1-ALK rearranged lung cancer. Cold Spring Harb Mol Case Stud. 2019 Nov 21. pii: mcs.a004630. doi: 10.1101/mcs.a004630. [Epub ahead of print]
- Kazdal D, Endris V, Allgäuer M, Kriegsmann M, Leichsenring J, Volckmar AL, Harms A, Kirchner M, Kriegsmann K, Neumann O, Brandt R, Talla SB, Rempel E, Ploeger C, von Winterfeld M, Christopoulos P, Merino DM, Stewart M, Allen J, Bischoff H, Meister M, Muley T, Herth F, Penzel R, Warth A, Winter H, Fröhling S, Peters S, Swanton C, Thomas M, Schirmacher P, Budczies J, Stenzinger A.Spatial and Temporal Heterogeneity of Panel-Based Tumor Mutational Burden in Pulmonary Adenocarcinoma: Separating Biology From Technical Artifacts.J Thorac Oncol. 2019 Nov;14(11):1935-1947. doi: 10.1016/j.jtho.2019.07.006. Epub 2019 Jul 23.
- Thielmann CM, Costa da Silva M, Muley T, Meister M, Herpel E, Muckenthaler MU. Iron accumulation in tumor-associated macrophages marks an improved overall survival in patients with lung adenocarcinoma. Sci Rep. 2019 Aug 5;9(1):11326. doi: 10.1038/s41598-019-47833-x.
- Ercetin E, Richtmann S, Delgado BM, Gomez-Mariano G, Wrenger S, Korenbaum E, Liu B, DeLuca D, Kühnel MP, Jonigk D, Yuskaeva K, Warth A, Muley T, Winter H, Meister M, Welte T, Janciauskiene S, Schneider MA. Clinical Significance of SERPINA1 Gene and Its Encoded Alpha1-antitrypsin Protein in NSCLC. Cancers (Basel). 2019 Sep 4;11(9). pii: E1306. doi: 10.3390/cancers11091306.
- Eichhorn F, Klotz LV, Muley T, Kobinger S, Winter H, Eichhorn ME. Prognostic relevance of regional lymph-node distribution in patients with N1-positive non-small cell lung cancer: A retrospective single-center analysis. Lung Cancer. 2019 Oct 20;138:95-101. doi: 10.1016/j.lungcan.2019.10.018. [Epub ahead of print
- Richtmann S, Wilkens D, Warth A, Lasitschka F, Winter H, Christopoulos P, Herth FJF, Muley T, Meister M, Schneider MA. FAM83A and FAM83B as Prognostic Biomarkers and Potential New Therapeutic Targets in NSCLC. Cancers (Basel). 2019 May 11;11(5). pii: E652. doi: 10.3390/cancers11050652
- Högerle BA, Lasitschka F, Muley T, Bougatf N, Herfarth K, Adeberg S, Eichhorn M, Debus J, Winter H, Rieken S, Uhl M. Primary adenoid cystic carcinoma of the trachea: clinical outcome of 38 patients after interdisciplinary treatment in a single institution. Radiat Oncol. 2019 Jul 4;14(1):117. doi: 10.1186/s13014-019-1323-
- Dabral S, Muecke C, Valasarajan C, Schmoranzer M, Wietelmann A, Semenza GL, Meister M, Muley T, Seeger-Nukpezah T, Samakovlis C, Weissmann N, Grimminger F, Seeger W, Savai R, Pullamsetti SS. A RASSF1A-HIF1α loop drives Warburg effect in cancer and pulmonary hypertension. Nat Commun. 2019 May 13;10(1):2130. doi: 10.1038/s41467-019-10044-z
- Muley T, He Y, Rolny V, Wehnl B, Escherich A, Warth A, Stolp C, Schneider MA, Meister M, Herth FJ, Dayyani F. Potential for the blood-based biomarkers cytokeratin 19 fragment (CYFRA 21-1) and human epididymal protein 4 (HE4) to detect recurrence during monitoring after surgical resection of adenocarcinoma of the lung. Lung Cancer. 2019 Apr;130:194-200. doi: 10.1016/j.lungcan.2019.02.017. Epub 2019 Feb 19.
- Li Y, Xiao X, Bossé Y, Gorlova O, Gorlov I, Han Y, Byun J, Leighl N, Johansen JS, Barnett M, Chen C, Goodman G, Cox A, Taylor F, Woll P, Wichmann HE, Manz J, Muley T, Risch A, Rosenberger , Han J, Siminovitch K, Arnold SM, Haura EB, Bolca C, Holcatova I, Janout V, Kontic M, Lissowska J, Mukeria A, Ognjanovic S, Orlowski TM, Scelo G, Swiatkowska B, Zaridze D, Bakke P, Skaug V, Zienolddiny S, Duell EJ, Butler LM, Houlston R, Artigas MS, Grankvist K, Johansson M, Shepherd FA, Marcus MW, Brunnström H, Manjer J, Melander O, Muller DC, Overvad K, Trichopoulou A, Tumino R, Liu G, Bojesen SE, Wu X, Le Marchand L, Albanes D, Bickeböller H, Aldrich MC, Bush WS, Tardon A, Rennert G, Teare MD, Field JK, Kiemeney LA, Lazarus P, Haugen A, Lam S, Schabath MB, Andrew AS, Bertazzi PA, Pesatori AC, Christiani DC, Caporaso N, Johansson M, McKay JD, Brennan P, Hung RJ, Amos CI. Genetic interaction analysis among oncogenesis-related genes revealed novel genes and networks in lung cancer development. Oncotarget. 2019 Mar 5;10(19):1760-1774. doi: 10.18632/oncotarget.26678. eCollection 2019 Mar 5.
- Hung RJ, Spitz MR, Houlston RS, Schwartz AG, Field JK, Ying J, Li Y, Han Y, Ji X, Chen W, Wu X, Gorlov IP, Na J, de Andrade M, Liu G, Brhane Y, Diao N, Wenzlaff A, Davies MPA, Liloglou T, Timofeeva M, Muley T, Rennert H, Saliba W, Ryan BM, Bowman E, Barros-Dios JM, Pérez-Ríos M, Morgenstern H, Zienolddiny S, Skaug V, Ugolini D, Bonassi S, van der Heijden EHFM, Tardon A, Bojesen SE, Landi MT, Johansson M, Bickeböller H, Arnold S, Le Marchand L, Melander O, Andrew A, Grankvist K, Caporaso N, Teare MD, Schabath MB, Aldrich MC, Kiemeney LA, Wichmann HE, Lazarus P, Mayordomo J, Neri M, Haugen A, Zhang ZF, Ruano-Raviña A, Brenner H, Harris CC, Orlow I, Rennert G, Risch A, Brennan P, Christiani DC, Amos CI, Yang P, Gorlova OY. Lung Cancer Risk in Never Smokers of European Descent is Associated with Genetic Variation in the 5P15.33 TERT-CLPTM1L Region. J Thorac Oncol. 2019 Apr 19. pii: S1556-0864(19)30289-8. doi: 10.1016/j.jtho.2019.04.008. [Epub ahead of print]
- Keith M. Kerr, Erik Thunnissen, Urania Dafnic, Stephen P. Finn, Lukas Bubendorf, Alex Soltermann, Eric Verbeken, Wojciech Biernat, Arne Warth, Antonio Marchetti, Ernst-Jan M. Speel, Sarawati Pokharel, Anne Marie Quinn, Kim Monkhorst, Atilio Navarro, Line Bille Madsen, Teodora Radonic, Joan Wilson, Graziano De Luca, Steven G. Gray, Richard Cheney, Spasenija Savic, Miguel Martorell, Thomas Muley, Paul Baas, Peter Meldgaard, Fiona Blackhall, Anne-Marie Dingemans, Rafal Dziadziuszko, Johan Vansteenkiste, Walter Weder, Varvara Polydoropoulou, Thomas Geiger, Roswitha Kammler, Solange Peters, Rolf Stahel, for the Lungscape Consortium. A retrospective cohort study of PD-L1 prevalence, molecular associations and clinical outcomes in patients with NSCLC: Results from the EuropeanThoracic Oncology Platform (ETOP) Lungscape Project Lung Cancer 2019, 131:95-103
- Kriegsmann K, Cremer M, Zgorzelski C, Harms A, Muley T, Winter H, Kazdal D, Warth A, Kriegsmann M. Agreement of CK5/6, p40, and p63 immunoreactivity in non-small cell lung cancer.Pathology. 2019 Feb 21. pii: S0031-3025(18)30490-2.doi: 0.1016/j.pathol.2018.11.009. [Epub ahead of print]
- Zhu Y, Wei Y, Zhang R, Dong X, Shen S, Zhao Y, Bai J, Albanes D, Caporaso NE, Landi MT, Zhu B, Chanock SJ, Gu F, Lam S, Tsao MS, Shepherd FA, Tardon A, Fernández-Somoano A, Fernandez-Tardon G, Chen C, Barnett MJ, Doherty J, Bojesen SE, Johansson M, Brennan P, McKay JD, Carreras-Torres R, Muley T, Risch A, Wichmann HE, Bickeboeller H, Rosenberger A, Rennert G, Saliba W, Arnold SM, Field JK, Davies MPA, Marcus MW, Wu X, Ye Y, Le Marchand L, Wilkens LR, Melander O, Manjer J, Brunnström H, Hung RJ, Liu G, Brhane Y, Kachuri L, Andrew AS, Duell EJ, Kiemeney LA, van der Heijden EHFM, Haugen A, Zienolddiny S, Skaug V, Grankvist K, Johansson M, Woll PJ, Cox A, Taylor F, Teare DM, Lazarus P, Schabath MB, Aldrich MC, Houlston RS, McLaughlin J, Stevens VL, Shen H, Hu Z, Dai J, Amos CI, Han Y, Zhu D, Goodman GE, Chen F, Christiani DC. Elevated platelet count appears to be causally associated with increased risk of lung cancer: A Mendelian randomization analysis. Cancer Epidemiol Biomarkers Prev. 2019 Jan 30. pii: cebp.0356.2018. doi: 10.1158/1055-9965.EPI-18-0356. [Epub ahead of print]
- Bains S, Eguchi T, Warth A, Yeh YC, Nitadori JI, Woo KM, Chou TY, Dienemann H, Muley T, Nakajima J, Shinozaki-Ushiku A, Wu YC, Lu S, Kadota K, Jones DR, Travis WD, Tan KS, Adusumilli PS. Procedure-Specific Risk Prediction for Recurrence in Patients Undergoing Lobectomy or Sublobar Resection for Small (≤2 cm) Lung Adenocarcinoma: An International Cohort Analysis. J Thorac Oncol. 2019 , 14: 72.86
- Christopoulos P, Kirchner M, Bozorgmehr F, Endris V, Elsayed M, Budczies J, Ristau J, Penzel R, Herth FJ, Heussel CP, Eichhorn M, Muley T, Meister M, Fischer JR, Rieken S, Lasitschka F, Bischoff H, Sotillo R, Schirmacher P, Thomas M, Stenzinger A. Identification of a highly lethal V3+ TP53+ subset in ALK+ lung adenocarcinoma. Int J Cancer. 2019; 144:190-199 doi: 10.1002/ijc.31893. [Epub ahead of print]
- Weber R, Meister M, Muley T, Thomas M, Sültmann H, Warth A, Winter H, Herth FJF, Schneider MA. Pathways regulating the expression of the immunomodulatory protein glycodelin in non‑small cell lung cancer. Int J Oncol. 2019 Feb;54(2):515-526. doi: 10.3892/ijo.2018.4654. Epub 2018 Dec 5.
2018
- Kazdal D, Harms A, Endris V, Penzel R, Oliveira C, Kriegsmann M, Longuespée R, Winter H, Schneider MA, Muley T, Pfarr N, Weichert W, Stenzinger A, Warth A. Subclonal evolution of pulmonary adenocarcinomas delineated by spatially distributed somatic mitochondrial mutations. Lung Cancer. 2018 Dec;126:80-88. doi: 10.1016/j.lungcan.2018.10.024. Epub 2018 Oct 29
- Roth A, Boulay K, Groß M, Polycarpou-Schwarz M, Mallette FA, Regnier M, Bida O, Ginsberg D, Warth A, Schnabel PA, Muley T, Meister M, Zabeck H, Hoffmann H, Diederichs S.Restoring LINC00673 expression triggers cellular senescence in lung cancer.RNA Biol. 2018 Nov 30. doi: 10.1080/15476286.2018.1553481. [Epub ahead of print]
- Marc A. Schneider, Thomas Muley, Rebecca Weber, Sabine Wessels, Michael Thomas, Felix J. F. Herth, Nicolas C. Kahn, Ralf Eberhardt, Hauke Winter, Gudula Heussel, Arne Warth, Christel Herold-Mende and Michael Meister. Glycodelin as a Serum and Tissue Biomarker for Metastatic and Advanced NSCLC Cancers 2018, 10(12), 486; doi: 10.3390/cancers10120486
- Thomas Muley, Vinzent Rolny, Ying He, Birgit Wehnl, Achim Escherich, Arne Warth, Christa Stolp, Marc A. Schneider, Hendrik Dienemann, Michael Meister, Felix J. Herth, Farshid Dayyani. The Combination of the Blood Based Tumor Biomarkers Cytokeratin 19 Fragments (CYFRA 21-1) and Carcinoembryonic Antigen (CEA) as a Potential Predictor of Benefit from Adjuvant Chemotherapy in Early Stage Squamous Cell Carcinoma of the Lung (SCC) Lung Cancer 2018, 120: 46-53
- Safi S, Op den Winkel J, Kramer S, Keast T, Wibowo H, Muley T, Herth FJF. A new bronchoscopic catheter for the transbronchial ablation of pulmonary nodules. Lung Cancer. 2018 Oct;124:125-129.
- Zabeck H, Dienemann H, Hoffmann H, Pfannschmidt J, Warth A, Schnabel PA, Muley T, Meister M, Sültmann H, Fröhlich H, Kuner R, Lasitschka F. Molecular signatures in IASLC/ATS/ERS classified growth patterns of lung adenocarcinoma. PLoS One. 2018 Oct 23;13(10):e0206132. doi: 10.1371/journal.pone.0206132. eCollection 2018
- Kriegsmann K, Zgorzelski C, Kazdal D, Cremer M, Muley T, Winter H, Longuespée R, Kriegsmann J, Warth A, Kriegsmann M. Insulinoma-associated Protein 1 (INSM1) in Thoracic Tumors is Less Sensitive but More Specific Compared With Synaptophysin, Chromogranin A, and CD56. Appl Immunohistochem Mol Morphol. 2018 Oct 24. doi: 10.1097/PAI.0000000000000715. [Epub ahead of print]
- Kriegsmann M, Kriegsmann K, Harms A, Longuespée R, Zgorzelski C, Leichsenring J, Muley T, Winter H, Kazdal D, Goeppert B, Warth A. Expression of HMB45, MelanA and SOX10 is rare in non-small cell lung cancer. Diagn Pathol. 2018 Sep 11;13(1):68. doi: 10.1186/s13000-018-0751-7.
- Ji X, Bossé Y, Landi MT, Gui J, Xiao X, Qian D, Joubert P, Lamontagne M, Li Y, Gorlov I, de Biasi M, Han Y, Gorlova O, Hung RJ, Wu X, McKay J, Zong X, Carreras-Torres R, Christiani DC, Caporaso N, Johansson M, Liu G, Bojesen SE, Le Marchand L, Albanes D, Bickeböller H, Aldrich MC, Bush WS, Tardon A, Rennert G, Chen C, Teare MD, Field JK, Kiemeney LA, Lazarus P, Haugen A, Lam S, Schabath MB, Andrew AS, Shen H, Hong YC, Yuan JM, Bertazzi PA, Pesatori AC, Ye Y, Diao N, Su L, Zhang R, Brhane Y, Leighl N, Johansen JS, Mellemgaard A, Saliba W, Haiman C, Wilkens L, Fernandez-Somoano A, Fernandez-Tardon G, van der Heijden EHFM, Kim JH, Dai J, Hu Z, Davies MPA, Marcus MW, Brunnström H, Manjer J, Melander O, Muller DC, Overvad K, Trichopoulou A, Tumino R, Doherty J, Goodman GE, Cox A, Taylor F, Woll P, Brüske I, Manz J, Muley T, Risch A, Rosenberger A, Grankvist K, Johansson M, Shepherd F, Tsao MS, Arnold SM, Haura EB, Bolca C, Holcatova I, Janout V, Kontic M, Lissowska J, Mukeria A, Ognjanovic S, Orlowski TM, Scelo G, Swiatkowska B, Zaridze D, Bakke P, Skaug V, Zienolddiny S, Duell EJ, Butler LM, Koh WP, Gao YT, Houlston R, McLaughlin J, Stevens V, Nickle DC, Obeidat M, Timens W, Zhu B, Song L, Artigas MS, Tobin MD, Wain LV, Gu F, Byun J, Kamal A, Zhu D, Tyndale RF, Wei WQ, Chanock S, Brennan P, Amos CI. Identification of susceptibility pathways for the role of chromosome 15q25.1 in modifying lung cancer risk.
- Nat Commun. 2018 Aug 13;9(1):3221. doi: 10.1038/s41467-018-05074-y.
- Christopoulos P, Chung I, Bozorgmehr F, Muley T, Meister M, Kobinger S, Marx A, Thomas M, Winter H, Herpel E, Rieker RJ, Fisch P, Grosch H. Deficient CD247 expression is a typical histopathologic characteristic of thymomas with cortical features. Histopathology. 2018; 73:1040-1043 doi: 10.1111/his.13724. [Epub ahead of print]
- Dvornikov D, Schneider MA, Ohse S, Szczygieł M, Titkova I, Rosenblatt M, Muley T, Warth A, Herth FJ, Dienemann H, Thomas M, Timmer J, Schilling M, Busch H, Boerries M, Meister M, Klingmüller U. Expression ratio of the TGFβ-inducible gene MYO10 is prognostic for overall survival of squamous cell lung cancer patients and predicts chemotherapy response. Sci Rep. 2018 Jun 22;8(1):9517. doi: 10.1038/s41598-018-27912-1
- Rosenberger A, Hung RJ, Christiani DC, Caporaso NE, Liu G, Bojesen SE, Le Marchand L, Haiman CA, Albanes D, Aldrich MC, Tardon A, Fernández-Tardón G, Rennert G, Field JK, Kiemeney B, Lazarus P, Haugen A, Zienolddiny S, Lam S, Schabath MB, Andrew AS, Brunnsstöm H, Goodman GE, Doherty JA, Chen C, Teare MD, Wichmann HE, Manz J, Risch A, Muley TR, Johansson M, Brennan P, Landi MT, Amos CI, Pesch B, Johnen G, Brüning T, Bickeböller H, Gomolka M. Genetic modifiers of radon-induced lung cancer risk: a genome-wide interaction study in former uranium miners. Int Arch Occup Environ Health. 2018; 91:937-950 doi: 10.1007/s00420-018-1334-3. [Epub ahead of print]
- Melnik S, Dvornikov D, Müller-Decker K, Depner S, Stannek P, Meister M, Warth A, Thomas M, Muley T, Risch A, Plass C, Klingmüller U, Niehrs C, Glinka A. Cancer cell specific inhibition of Wnt/β-catenin signaling by forced intracellular acidification. Cell Discov. 2018 Jul 3;4:37. doi: 10.1038/s41421-018-0033-2. eCollection 2018
- Kahn N, Rossler AK, Hornemann K, Muley T, Grünig E, Schmidt W, Herth FJF, Kreuter M. C-proSP-B: A Possible Biomarker for Pulmonary Diseases? Respiration. 2018 May 15:1-10. doi: 10.1159/000488245.
- Christopoulos P, Schneider MA, Bozorgmehr F, Kuon J, Engel-Riedel W, Kollmeier J, Baum V, Muley T, Schnabel PA, Bischoff H, Grohé C, Serke M, Thomas M, Fisch P, Meister M.
Large cell neuroendocrine lung carcinoma induces peripheral T-cell repertoire alterations with predictive and prognostic significance. Lung Cancer. 2018 May;119:48-55. doi: 10.1016/j.lungcan.2018.03.002. Epub 2018 Mar 6. - Yamauchi Y, Safi S, Blattner C, Rathinasamy A, Umansky L, Juenger S, Warth A, Eichhorn M, Muley T, Herth FJF, Dienemann H, Platten M, Beckhove P, Utikal J, Hoffmann H, Umansky V Circulating and Tumor Myeloid-derived Suppressor Cells in Resectable Non-small-cell Lung Cancer. Am J Respir Crit Care Med. 2018; 198:777-787 Apr 4. doi: 10.1164/rccm.201708-1707OC. [Epub ahead of print]
- Eichhorn F, Harms A, Warth A, Muley T, Winter H, Eichhorn ME. PD-L1 expression in large cell neuroendocrine carcinoma of the lung. Lung Cancer. 2018 Apr; 118:76-82.
- Kriegsmann M, Harms A, Kazdal D, Fischer S, Stenzinger A, Leichsenring J, Penzel R, Longuespée R, Kriegsmann K, Muley T, Safi S, Warth A. Analysis of the proliferative activity in lung adenocarcinomas with specific driver mutations. Pathol Res Pract. 2018 Mar;214(3):408-416.
- Kriegsmann M, Harms A, Longuespée R, Muley T, Winter H, Kriegsmann K, Kazdal D, Goeppert B, Pathil A, Warth A. Role of conventional immunomarkers, HNF4-α and SATB2, in the differential diagnosis of pulmonary and colorectal adenocarcinomas. Histopathology. 2018 May;72(6):997-1006. doi: 10.1111/his.13455. Epub 2018 Feb 20
- Radovich M, Pickering CR, Felau 3, Ha G, Zhang H, Jo H, Hoadley KA, Anur P, Zhang J, McLellan M, Bowlby R, Matthew T, Danilova L, Hegde AM, Kim J, Leiserson MDM, Sethi G, Lu C, Ryan M, Su X, Cherniack AD, Robertson G, Akbani R, Spellman P, Weinstein JN, Hayes DN, Raphael B, Lichtenberg T, Leraas K, Zenklusen JC; Cancer Genome Atlas Network, Fujimoto J, Scapulatempo-Neto C, Moreira AL, Hwang D, Huang J, Marino M, Korst R, Giaccone G, Gokmen-Polar Y, Badve S, Rajan A, Ströbel P, Girard N, Tsao MS, Marx A, Tsao AS, Loehrer PJ.Collaborators (141)Ally A, Appelbaum EL, Auman JT, Balasundaram M, Balu S, Behera M, Beroukhim R, Berrios M, Blandino G, Bodenheimer T, Bootwalla MS, Bowen J, Brooks D, Carcano FM, Carlsen R, Carvalho AL, Castro P, Chalabreysse L, Chin L, Cho J, Choe G, Chuah E, Chudamani S, Cibulskis C, Cope L, Cordes MG, Crain D, Curley E, Defreitas T, Demchok JA, Detterbeck F, Dhalla N, Dienemann H, Edenfield WJ, Facciolo F, Ferguson ML, Frazer S, Fronick CC, Fulton LA, Fulton RS, Gabriel SB, Gardner J, Gastier-Foster JM, Gehlenborg N, Gerken M, Getz G, Heiman DI, Hobensack S, Holbrook A, Holt RA, Hoyle AP, Hutter CM, Ittmann M, Jefferys SR, Jones CD, Jones SJM, Kasaian K, Kim J, Kimes PK, Lai PH, Laird PW, Lawrence MS, Lin P, Liu J, Lolla L, Lu Y, Ma Y, Maglinte DT, Mallery D, Mardis ER, Marra MA, Martin J, Mayo M, Meier S, Meister M, Meng S, Meyerson M, Mieczkowski PA, Miller CA, Mills GB, Moore RA, Morris S, Mose LE, Muley T, Mungall AJ, Mungall K, Naresh R, Newton Y, Noble MS, Owonikoko T, Parker JS, Paulaskis J, Penny R, Perou CM, Perrin C, Pihl T, Radenbaugh A, Ramalingam S, Ramirez N, Rieker R, Roach J, Sadeghi S, Saksena G, Schein JE, Schmidt HK, Schumacher SE, Shelton C, Shelton T, Shi Y, Shih J, Sica G, Silveira HCS, Simons JV, Sipahimalani P, Skelly T, Sofia HJ, Soloway MG, Stuart J, Sun Q, Tam A, Tan D, Tarnuzzer R, Thiessen N, Van Den Berg DJ, Vasef MA, Veluvolu U, Voet D, Walter V, Wan Y, Wang Z, Warth A, Weis CA, Weisenberger DJ, Wilkerson MD, Wise L, Wong T, Wu HT, Wu Y, Yang L, Zhang J, Zmuda E. The Integrated Genomic Landscape of Thymic Epithelial Tumors. Cancer Cell. 2018 Feb 12;33(2):244-258.e10. doi: 10.1016/j.ccell.2018.01.003.
- Gonzalez-Vallinas M, Rodriguez-Paredes M, Albrecht M, Sticht C, Stichel D, Gutekunst J, Pitea A, Sass S, Sánchez-Rivera FJ, Bermejo JL, Schmitt J, De La Torre C, Warth A, Theis F, Mueller N, Gretz N, Muley T, Meister M, Tschaharganeh DF, Schirmacher P, Matthäus F, Breuhahn K. Epigenetically Regulated Chromosome 14q32 miRNA Cluster Induces Metastasis and Predicts Poor Prognosis in Lung Adenocarcinoma Patients. Mol Cancer Res. 2018; 16:390-402 pii: molcanres.0334.2017. doi: 10.1158/1541-7786.MCR-17-0334. [Epub ahead of print]
- Christopoulos P, Endris V, Bozorgmehr F, Elsayed M, Kirchner M, Ristau J, Buchhalter I, Penzel R, Herth FJ, Heussel CP, Eichhorn M, Muley T, Meister M, Fischer JR, Rieken S, Warth A, Bischoff H, Schirmacher P, Stenzinger A, Thomas M. EML4-ALK fusion variant V3 is a high-risk feature conferring accelerated metastatic spread, early treatment failure and worse overall survival in ALK(+). non-small cell lung cancer. Int J Cancer. 2018; 142:2589-2598 doi: 10.1002/ijc.31275. [Epub ahead of print] PubMed PMID: 29363116.
- Letovanec I, Finn S, Zygoura P, Smyth P, Soltermann A, Bubendorf L, Speel EJ, Marchetti A, Nonaka D, Monkhorst K, Hager H, Martorell M, Sejda A, Cheney R, Hernandez-Losa J, Verbeken E, Weder W, Savic S, Di Lorito A, Navarro A, Felip E, Warth A, Baas P, Meldgaard P, Blackhall F, Dingemans AM, Dienemann H, Dziadziuszko R, Vansteenkiste J, O'Brien C, Geiger T, Sherlock J, Schageman J, Dafni U, Kammler R, Kerr K, Thunnissen E, Stahel R, Peters S; European Thoracic Oncology Platform Lungscape Consortium. Evaluation of NGS and RT-PCR Methods for ALK Rearrangement in European NSCLC Patients: Results from the European Thoracic Oncology Platform Lungscape Project. J Thorac Oncol. 2018 Mar;13(3):413-425. doi: 10.1016/j.jtho.2017.11.117. Epub 2017 Nov 27. PubMed PMID: 29191776.
- Kerr KM, Dafni U, Schulze K, Thunnissen E, Bubendorf L, Hager H, Finn S, Biernat W, Vliegen L, Losa JH, Marchetti A, Cheney R, Warth A, Speel EJ, Blackhall F, Monkhorst K, Jantus Lewintre E, Tischler V, Clark C, Bertran-Alamillo J, Meldgaard P, Gately K, Wrona A, Vandenberghe P, Felip E, De Luca G, Savic S, Muley T, Smit EF, Dingemans AC, Priest L, Baas P, Camps C, Weder W, Polydoropoulou V, Geiger TR, Kammler R, Sumiyoshi T, Molina MA, Shames DS, Stahel RA, Peters S; ETOP Lungscape Consortium. Prevalence and clinical association of gene mutations through multiplex mutation testing in patients with NSCLC: results from the ETOP Lungscape Project. Ann Oncol. 2018 Jan 1;29(1):200-208. doi: 10.1093/annonc/mdx629. PubMed PMID: 29186353.
2017
- Yamauchi Y, Safi S, Muley T, Warth A, Herth FJF, Dienemann H, Hoffmann H, Eichhorn ME. C-reactive protein-albumin ratio is an independent prognostic predictor of tumor recurrence in stage IIIA-N2 lung adenocarcinoma patients. Lung Cancer. 2017 Dec;114:62-67. doi: 10.1016/j.lungcan.2017.11.002. Epub 2017 Nov 3. PubMed PMID: 29173768.
- Kriegsmann M, Harms A, Longuespée R, Muley T, Winter H, Kriegsmann K, Kazdal D, Goeppert B, Pathil A, Warth A. Role of Conventional Immunomarkers, HNF4-a, and SATB2 in the Differential Diagnosis of Pulmonary and Colorectal Adenocarcinomas. Histopathology. 2017 Dec 15. doi: 10.1111/his.13455. [Epub ahead of print]
- Li Y, Xiao X, Han Y, Gorlova O, Qian D, Leighl N, Johansen JS, Barnett M, Chen C, Goodman G, Cox A, Taylor F, Woll P, Wichmann HE, Manz J, Muley T, Risch A, Rosenberger A, Arnold SM, Haura EB, Bolca C, Holcatova I, Janout V, Kontic M, Lissowska J, Mukeria A, Ognjanovic S, Orlowski TM, Scelo G, Swiatkowska B, Zaridze D, Bakke P, Skaug V, Zienolddiny S, Duell EJ, Butler LM, Houlston R, Artigas MS, Grankvist K, Johansson M, Shepherd FA, Marcus MW, Brunnström H, Manjer J, Melander O, Muller DC, Overvad K, Trichopoulou A, Tumino R, Liu G, Bojesen SE, Wu X, Le Marchand L, Albanes D, Bickeböller H, Aldrich MC, Bush WS, Tardon A, Rennert G, Teare MD, Field JK, Kiemeney LA, Lazarus P, Haugen A, Lam S, Schabath MB, Andrew AS, Bertazzi PA, Pesatori AC, Christiani DC, Caporaso N, Johansson M, McKay JD, Brennan P, Hung RJ, Amos CI. Genome-wide interaction study of smoking behavior and non-small cell lung cancer risk in Caucasian population. Carcinogenesis. 2017 Oct 20. doi: 10.1093/carcin/bgx113. [Epub ahead of print
- Schmidt LH, Stucke-Ring J, Brand C, Schliemann C, Harrach S, Muley T, Herpel E, Kessler T, Mohr M, Görlich D, Kreuter M, Lenz G, Wardelmann E, Thomas M, Berdel WE, Schwöppe C, Hartmann W. CD13 as target for tissue factor induced tumor vascular infarction in small cell lung cancer.Lung Cancer. 2017 Nov;113:121-127. doi: 10.1016/j.lungcan.2017.09.013. Epub 2017 Sep 22.
- Costa da Silva M, Breckwoldt MO, Vinchi F, Correia MP, Stojanovic A, Thielmann CM, Meister M, Muley T, Warth A, Platten M, Hentze MW, Cerwenka A, Muckenthaler MU. Iron Induces Anti-tumor Activity in Tumor-Associated Macrophages. Front Immunol. 2017 Nov 8;8:1479. doi: 10.3389/fimmu.2017.01479. eCollection 2017
- K.M. Kerr, U. Dafni, K. Schulze, E. Thunnissen, L. Bubendorf, H. Hager, S. Finn, W. Biernat, L. Vliegen, J. H. Losa, A. Marchetti, R. Cheney, A. Warth, E-J. Speel, F. Blackhall, K. Monkhorst, E. Jantus Lewintre, V. Tischler, C. Clark, J. Bertran-Alamillo, P. Meldgaard, K. Gately, A. Wrona, P. Vandenberghe, E. Felip, G. De Luca, S. Savic, T. Muley, E. F. Smit, A-M. C. Dingemans, L. Priest, P. Baas, C. Camps, W. Weder, V. Polydoropoulou, T.R. Geiger, R. Kammler, T. Sumiyoshi, M. A. Molina, D. S. Shames, R.A. Stahel, S. Peters, for the ETOP Lungscape Consortium. Prevalence and clinical association of gene mutations through Multiplex Mutation Testing in patients with NSCLC: Results from the ETOP Lungscape Project Annals of Oncology 2017, MDX629
- Zhao J, Dinkel J, Warth A, Penzel R, Reinmuth N, Schnabel P, Muley T, Meister M, Zabeck H, Steins M, Yang JY, Zhou Q, Schlemmer HP, Herth FJF, Kauczor HU, Heussel CP. CT characteristics in pulmonary adenocarcinoma with epidermal growth factor receptor mutation. PLoS One. 2017 Sep 26;12(9):e0182741. doi: 10.1371/journal.pone.0182741. eCollection 2017
- Kazdal D, Harms A, Endris V, Penzel R, Kriegsmann M, Eichhorn F, Muley T, Stenzinger A, Pfarr N, Weichert W, Warth A. Prevalence of somatic mitochondrial mutations and spatial distribution of mitochondria in non-small cell lung cancer. Br J Cancer. 2017 May 30. doi: 10.1038/bjc.2017.155. [Epub ahead of print]
- McKay JD, Hung RJ, Han Y, Zong X, Carreras-Torres R, Christiani DC, Caporaso NE, Johansson M, Xiao X, Li Y, Byun J, Dunning A, Pooley KA, Qian DC, Ji X, Liu G, Timofeeva MN, Bojesen SE, Wu X, Le Marchand L, Albanes D, Bickeböller H, Aldrich MC, Bush WS, Tardon A, Rennert G, Teare MD, Field JK, Kiemeney LA, Lazarus P, Haugen A, Lam S, Schabath MB, Andrew AS, Shen H, Hong YC, Yuan JM, Bertazzi PA, Pesatori AC, Ye Y, Diao N, Su L, Zhang R, Brhane Y, Leighl N, Johansen JS, Mellemgaard A, Saliba W, Haiman CA, Wilkens LR, Fernandez-Somoano A, Fernandez-Tardon G, van der Heijden HFM, Kim JH, Dai J, Hu Z, Davies MPA, Marcus MW, Brunnström H, Manjer J, Melander O, Muller DC, Overvad K, Trichopoulou A, Tumino R, Doherty JA, Barnett MP, Chen C, Goodman GE, Cox A, Taylor F, Woll P, Brüske I, Wichmann HE, Manz J, Muley TR, Risch A, Rosenberger A, Grankvist K, Johansson M, Shepherd FA, Tsao MS, Arnold SM, Haura EB, Bolca C, Holcatova I, Janout V, Kontic M, Lissowska J, Mukeria A, Ognjanovic S, Orlowski TM, Scelo G, Swiatkowska B, Zaridze D, Bakke P, Skaug V, Zienolddiny S, Duell EJ, Butler LM, Koh WP, Gao YT, Houlston RS, McLaughlin J, Stevens VL, Joubert P, Lamontagne M, Nickle DC, Obeidat M, Timens W, Zhu B, Song L, Kachuri L, Artigas MS, Tobin MD, Wain LV; SpiroMeta Consortium, Rafnar T, Thorgeirsson TE, Reginsson GW, Stefansson K, Hancock DB, Bierut LJ, Spitz MR, Gaddis NC, Lutz SM, Gu F, Johnson EO, Kamal A, Pikielny C, Zhu D, Lindströem S, Jiang X, Tyndale RF, Chenevix-Trench G, Beesley J, Bossé Y, Chanock S, Brennan P, Landi MT, Amos CI. Large-scale association analysis identifies new lung cancer susceptibility loci and heterogeneity in genetic susceptibility across histological subtypes. Nat Genet. 2017 Jun 12. doi: 10.1038/ng.3892. [Epub ahead of print]
- Gautschi O, Milia J, Filleron T, Wolf J, Carbone DP, Owen D, Camidge R, Narayanan V, Doebele RC, Besse B, Remon-Masip J, Janne PA, Awad MM, Peled N, Byoung CC, Karp DD, Van Den Heuvel M, Wakelee HA, Neal JW, Mok TSK, Yang JCH, Ou SI, Pall G, Froesch P, Zalcman G, Gandara DR, Riess JW, Velcheti V, Zeidler K, Diebold J, Früh M, Michels S, Monnet I, Popat S, Rosell R, Karachaliou N, Rothschild SI, Shih JY, Warth A, Muley T, Cabillic F, Mazières J, Drilon A. Targeting RET in Patients With RET-Rearranged Lung Cancers: Results From the Global, Multicenter RET Registry. J Clin Oncol. 2017 May 1;35(13):1403-1410. doi: 10.1200/JCO.2016.70.9352. Epub 2017 Mar 13.
- Guo M, Tomoshige K, Meister M, Muley T, Fukazawa T, Tsuchiya T, Karns R, Warth A, Fink-Baldauf IM, Nagayasu T, Naomoto Y, Xu Y, Mall MA, Maeda Y. Gene signature driving invasive mucinous adenocarcinoma of the lung. EMBO Mol Med. 2017 Mar 2. pii: e201606711. doi: 10.15252/emmm.201606711. [Epub ahead of print]
- Nattenmüller J, Wochner R, Muley T, Steins M, Hummler S, Teucher B, Wiskemann J, Kauczor HU, Wielpütz MO, Heussel CP. Prognostic Impact of CT-Quantified Muscle and Fat Distribution before and after First-Line-Chemotherapy in Lung Cancer Patients. PLoS One. 2017 Jan 20;12(1):e0169136. doi: 10.1371/journal.pone.0169136.
- Schneider MA, Christopoulos P, Muley T, Warth A, Klingmueller U, Thomas M, Herth FJ, Dienemann H, Mueller NS, Theis F, Meister M. AURKA, DLGAP5, TPX2, KIF11 and CKAP5: Five specific mitosis-associated genes correlate with poor prognosis for non-small cell lung cancer patients. Int J Oncol. 2017 Feb;50(2):365-372. doi: 10.3892/ijo.2017.3834.
- Herpel E, Rieker RJ, Dienemann H, Muley T, Meister M, Hartmann A, Warth A, Agaimy A SMARCA4 and SMARCA2 deficiency in non-small cell lung cancer: immunohistochemical survey of 316 consecutive specimens. Ann Diagn Pathol. 2017 Feb;26:47-51.
2016
- Schneider MA, Muley T, Kahn NC, Warth A, Thomas M, Herth FJ, Dienemann H, Meister M. Glycodelin is a potential novel follow-up biomarker for malignant pleural mesothelioma. Oncotarget. 2016 Oct 4. doi: 10.18632/oncotarget.12474.
- Riediger AL, Dietz S, Schirmer U, Meister M, Heinzmann-Groth I, Schneider M, Muley T, Thomas M, Sültmann H. Mutation analysis of circulating plasma DNA to determine response to EGFR tyrosine kinase inhibitor therapy of lung adenocarcinoma patients. Sci Rep. 2016 Sep 19;6:33505. doi: 10.1038/srep33505.
- Firnkorn D, Merker S, Ganzinger M, Muley T, Knaup P.Unlocking Data for Statistical Analyses and Data Mining: Generic Case Extraction of Clinical Items from i2b2 and tranSMART. Stud Health Technol Inform. 2016;228:567-71
- Dietz S, Schirmer U, Mercé C, von Bubnoff N, Dahl E, Meister M, Muley T, Thomas M, Sültmann H. Low Input Whole-Exome Sequencing to Determine the Representation of the Tumor Exome in Circulating DNA of Non-Small Cell Lung Cancer Patients. PLoS One. 2016,11(8):e0161012.
- Campbell JD, Alexandrov A, Kim J, Wala J, Berger AH, Pedamallu CS, Shukla SA, Guo G, Brooks AN, Murray BA, Imielinski M, Hu X, Ling S, Akbani R, Rosenberg M, Cibulskis C, Ramachandran A, Collisson EA, Kwiatkowski DJ, Lawrence MS, Weinstein JN, Verhaak RG, Wu CJ, Hammerman PS, Cherniack AD, Getz G; Cancer Genome Atlas Research Network, Artyomov MN, Schreiber R, Govindan R, Meyerson M. Distinct patterns of somatic genome alterations in lung adenocarcinomas and squamous cell carcinomas. Nat Genet. 2016 Jun;48(6):607-16
- Warth A, Endris V, Stenzinger A, Penzel R, Harms A, Duell T, Abdollahi A, Lindner M, Schirmacher P, Muley T, Dienemann H, Fink L, Morresi-Hauf A, Pfarr N, Weichert W. Genetic changes of non-small cell lung cancer under neoadjuvant therapy. Oncotarget. 2016 Apr 20. doi: 10.18632/oncotarget.8858.
2015
- Firnkorn D, Ganzinger M, Muley T, Thomas M, Knaup P. A Generic Data Harmonization Process for Cross-linked Research and Network Interaction. Construction and Application for the Lung Cancer Phenotype Database of the German Center for Lung Research. Methods Inf Med. 2015, 54(5): 455-460.
- Brenner DR, Amos CI, Brhane Y, Timofeeva MN, Caporaso N, Wang Y, Christiani DC, Bickeböller H, Yang P, Albanes D, Stevens VL, Gapstur S, McKay J, Boffetta P, Zaridze D, Szeszenia-Dabrowska N, Lissowska J, Rudnai P, Fabianova E, Mates D, Bencko V, Foretova L, Janout V, Krokan HE, Skorpen F, Gabrielsen ME, Vatten L, Njølstad I, Chen C, Goodman G, Lathrop M, Vooder T, Välk K, Nelis M, Metspalu A, Broderick P, Eisen T, Wu X, Zhang D, Chen W, Spitz MR, Wei Y, Su L, Xie D, She J, Matsuo K, Matsuda F, Ito H, Risch A, Heinrich J, Rosenberger A, Muley T, Dienemann H, Field JK, Raji O, Chen Y, Gosney J, Liloglou T, Davies MP, Marcus M, McLaughlin J, Orlow I, Han Y, Li Y, Zong X, Johansson M; EPIC Investigators, Liu G, Tworoger SS, Le Marchand L, Henderson BE, Wilkens LR, Dai J, Shen H, Houlston RS, Landi MT, Brennan P, Hung RJ. Identification of lung cancer histology-specific variants applying Bayesian framework variant prioritization approaches within the TRICL and ILCCO consortia. Carcinogenesis. 2015 Sep 10. pii: bgv128. [Epub ahead of print]
- Kahn N, Riedlinger J, Roeßler M, Rabe C, Lindner M, Koch I, Schott-Hildebrand S, Herth FJ, Schneider A M, Meister M, Muley TR. Blood-sampling collection prior to surgery may have a significant influence upon biomarker concentrations measured. Clinical Proteomics.2015, 12:19; DOI: 10.1186/s12014-015-9093-6. URL: http://www.clinicalproteomicsjournal.com/content/12/1/19
- Schneider MA, Granzow M, Warth A, Schnabel PA, Thomas M, Herth FJ, Dienemann H, Muley T, Meister M. Glycodelin: A new Biomarker with Immunomodulatory Functions in Non-Small Cell Lung Cancer. Clin Cancer Res. 2015, 21(15):3529-40.